Toda a função biológica depende de como diferentes proteínas interagem entre si. As interações proteína-proteína facilitam tudo, desde transcrição do DNA e controle da divisão celular até funções de nível superior em organismos complexos.
Muito ainda não está claro sobre como essas funções são orquestradas no nível molecular e como as proteínas interagem entre si – seja com outras proteínas ou com cópias de si mesmas.
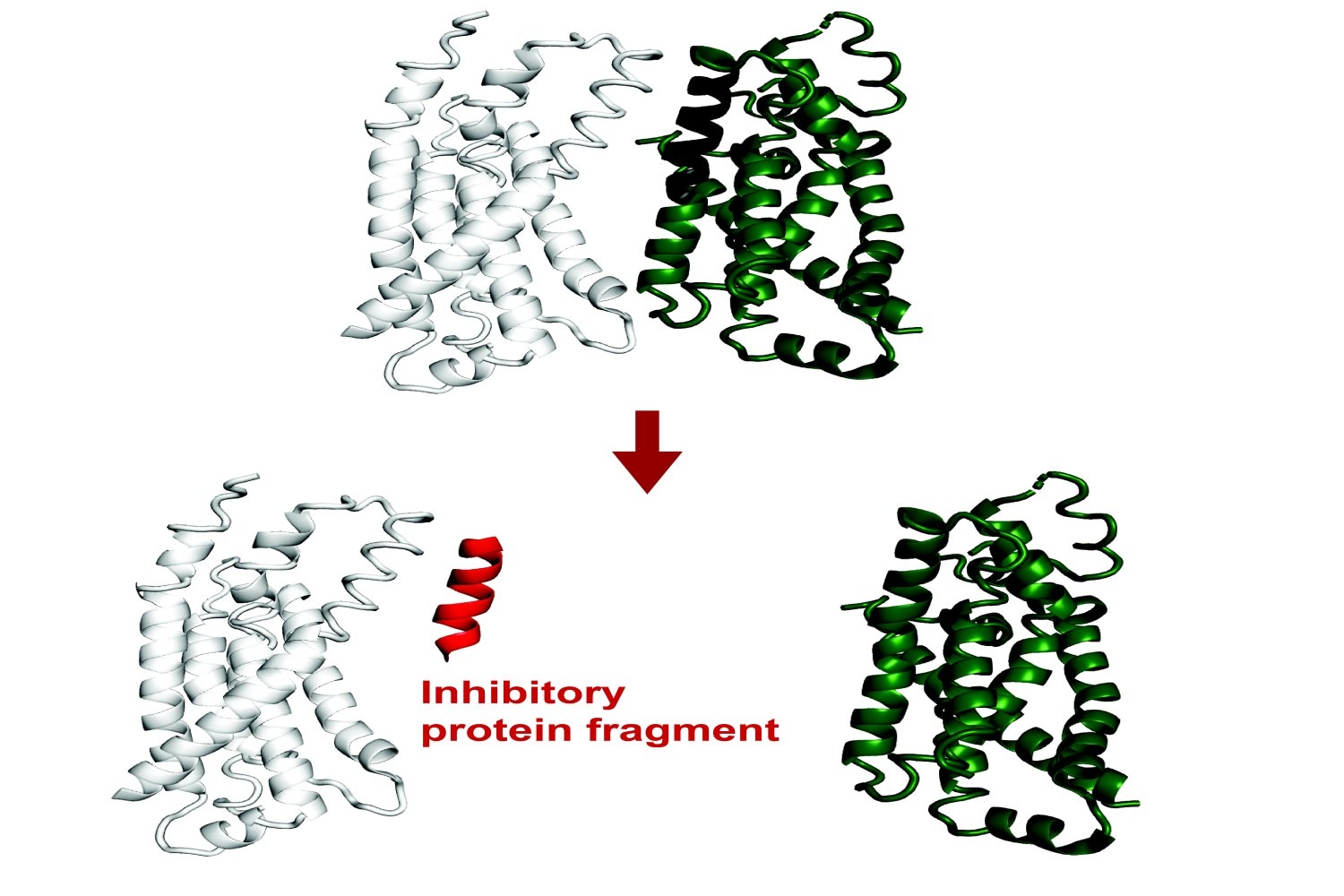
Achados recentes revelaram que pequenos fragmentos de proteína têm muito potencial funcional. Embora sejam peças incompletas, trechos curtos de aminoácidos ainda podem se ligar a interfaces de uma proteína alvo, recapitulando as interações nativas. Através desse processo, eles podem alterar a função dessa proteína ou interromper suas interações com outras proteínas.
Os fragmentos de proteínas poderiam, portanto, capacitar a pesquisa básica sobre interações proteicas e processos celulares e poderiam potencialmente ter aplicações terapêuticas.
Recentemente publicado em Anais da Academia Nacional de Ciênciasum novo método desenvolvido no Departamento de Biologia baseia-se nos modelos de inteligência synthetic existentes para prever computacionalmente fragmentos de proteínas que podem se ligar e inibir proteínas completas em E. coli. Teoricamente, essa ferramenta pode levar a inibidores geneticamente codificáveis contra qualquer proteína.
O trabalho foi realizado no laboratório de Professor Associado de Biologia e Investigador do Instituto Médico Howard Hughes Gene-Wei Li Em colaboração com o laboratório de Jay A. Stein (1968) Professor de Biologia, Professor de Engenharia Biológica e Chefe de Departamento Amy Keating.
Aproveitando o aprendizado de máquina
O programa, chamado FragFold, aproveita Alphafold, um modelo de IA que levou a avanços fenomenais na biologia nos últimos anos devido à sua capacidade de prever as interações dobráveis de proteínas e proteínas.
O objetivo do projeto period prever inibidores de fragmentos, que é uma nova aplicação de alfafold. Os pesquisadores deste projeto confirmaram experimentalmente que mais da metade das previsões de Fragfold para ligação ou inibição eram precisas, mesmo quando os pesquisadores não tinham dados estruturais anteriores sobre os mecanismos dessas interações.
“Nossos resultados sugerem que essa é uma abordagem generalizável para encontrar modos de ligação que provavelmente inibirão a função de proteína, inclusive para novos alvos de proteínas, e você pode usar essas previsões como ponto de partida para outras experiências”, diz co-primeiro e autor correspondente. Andrew Savinov, um pós -doutorado no laboratório Li. “Podemos realmente aplicar isso a proteínas sem funções conhecidas, sem interações conhecidas, sem estruturas conhecidas, e podemos colocar alguma credibilidade nesses modelos que estamos desenvolvendo”.
Um exemplo é o FTSZ, uma proteína que é basic para a divisão celular. Está bem estudado, mas contém uma região intrinsecamente desordenada e, portanto, especialmente desafiadora de estudar. As proteínas desordenadas são dinâmicas e suas interações funcionais são muito provavelmente fugazes – ocorrendo tão brevemente que as ferramentas atuais de biologia estrutural não podem capturar uma única estrutura ou interação.
Os pesquisadores aproveitaram o Fragfold para explorar a atividade de fragmentos de FTSZ, incluindo fragmentos da região intrinsecamente desordenada, para identificar várias novas interações de ligação com várias proteínas. Esse salto no entendimento confirma e expande os experimentos anteriores que medem a atividade biológica da FTSZ.
Esse progresso é significativo em parte porque foi feito sem resolver a estrutura da região desordenada e porque exibe o poder potencial do Fragfold.
“Este é um exemplo de como o Alphafold está mudando fundamentalmente como podemos estudar biologia molecular e celular”, diz Keating. “Aplicações criativas dos métodos de IA, como nosso trabalho em FragFold, abrem recursos inesperados e novas direções de pesquisa”.
Inibição e além
Os pesquisadores realizaram essas previsões fragmentando computacionalmente cada proteína e depois modelando como esses fragmentos se ligariam aos parceiros de interação que achavam relevante.
Eles compararam os mapas de ligação prevista em toda a sequência com os efeitos desses mesmos fragmentos nas células vivas, determinadas usando medições experimentais de alto rendimento nas quais milhões de células produzem um tipo de fragmento de proteína.
O Alphafold usa informações co-evolutivas para prever o dobramento e normalmente avalia a história evolutiva das proteínas usando algo chamado alinhamento de múltiplas seqüências para todas as previsões. Os MSAs são críticos, mas são um gargalo para previsões em larga escala-eles podem levar uma quantidade proibitiva de tempo e poder computacional.
Para o FragFold, os pesquisadores pré-calculam o MSA para uma proteína completa uma vez e usaram esse resultado para guiar as previsões para cada fragmento dessa proteína completa.
Savinov, juntamente com o ex -aluno do Keating Lab Sebastian Swanson PhD ’23, previu fragmentos inibitórios de um conjunto diversificado de proteínas, além da FTSZ. Entre as interações que eles exploraram estava um complexo entre as proteínas de transporte de lipopolissacarídeos LPTF e LPTG. Um fragmento de proteína de LPTG inibiu essa interação, presumivelmente interrompendo a entrega do lipopolissacarídeo, que é um componente essential do E. coli Membrana celular externa essencial para a aptidão celular.
“A grande surpresa foi que podemos prever a ligação com uma precisão tão alta e, de fato, geralmente preveja a ligação que corresponde à inibição”, diz Savinov. “Para todas as proteínas que analisamos, conseguimos encontrar inibidores”.
Os pesquisadores inicialmente se concentraram em fragmentos de proteínas como inibidores, porque se um fragmento pode bloquear uma função essencial nas células é um resultado relativamente simples para medir sistematicamente. Olhando para o futuro, Savinov também está interessado em explorar a função de fragmento externa, como fragmentos que podem estabilizar a proteína à qual se ligam, aprimoram ou alteram sua função ou a degradação de proteínas.
Design, em princípio
Esta pesquisa é um ponto de partida para o desenvolvimento de uma compreensão sistêmica dos princípios de design celular e quais elementos modelos de aprendizado profundo podem estar se baseando para fazer previsões precisas.
“Há uma meta mais ampla e de alcance que estamos construindo”, diz Savinov. “Agora que podemos prever, podemos usar os dados que temos de previsões e experimentos para extrair os recursos salientes para descobrir o que o Alphafold realmente aprendeu sobre o que faz um bom inibidor?”
Savinov e colaboradores também investigaram ainda mais como os fragmentos de proteínas se ligam, explorando outras interações proteicas e mutando resíduos específicos para ver como essas interações mudam a maneira como o fragmento interage com seu alvo.
Examinando experimentalmente o comportamento de milhares de fragmentos mutados nas células, uma abordagem conhecida como varredura mutacional profunda, revelou aminoácidos importantes responsáveis pela inibição. Em alguns casos, os fragmentos mutados eram inibidores ainda mais potentes do que suas seqüências naturais e completas.
“Ao contrário dos métodos anteriores, não nos limitamos a identificar fragmentos em dados estruturais experimentais”, diz Swanson. “A força central deste trabalho é a interação entre dados de inibição experimental de alta rendição e os modelos estruturais previstos: os dados experimentais nos guia para os fragmentos que são particularmente interessantes, enquanto os modelos estruturais previstos por fragsolos fornecem uma hipótese específica e testável para Como os fragmentos funcionam em um nível molecular. ”
Savinov está empolgado com o futuro dessa abordagem e suas inúmeras aplicações.
“Ao criar ligantes compactos e geneticamente codificáveis, o Fragfold abre uma ampla gama de possibilidades para manipular a função proteica”, concorda Li. “Podemos imaginar fornecer fragmentos funcionalizados que podem modificar proteínas nativas, alterar sua localização subcelular e até reprogramá -las para criar novas ferramentas para estudar a biologia celular e tratar doenças”.