Uma proteína localizada na parte errada de uma célula pode contribuir para várias doenças, como Alzheimer, fibrose cística e câncer. Mas existem cerca de 70.000 variantes diferentes de proteínas e proteínas em uma única célula humana e, como os cientistas normalmente podem testar apenas um punhado em um experimento, é extremamente caro e demorado para identificar manualmente os locais das proteínas.
Uma nova geração de técnicas computacionais busca otimizar o processo usando modelos de aprendizado de máquina que geralmente aproveitam os conjuntos de dados contendo milhares de proteínas e seus locais, medidos em várias linhas celulares. Um dos maiores conjuntos de dados é o atlas de proteínas humanas, que cataloga o comportamento subcelular de Over 13.000 proteínas em mais de 40 linhas celulares. Mas, por mais enorme que seja, o Atlas da Proteína Humana explorou apenas cerca de 0,25 % de todos os pares possíveis de todas as proteínas e linhas celulares dentro do banco de dados.
Agora, pesquisadores do MIT, Universidade de Harvard e do Broad Institute of MIT e Harvard desenvolveram uma nova abordagem computacional que pode explorar com eficiência o espaço restante desconhecido. Seu método pode prever a localização de qualquer proteína em qualquer linha celular humana, mesmo quando a proteína e a célula nunca foram testadas antes.
Sua técnica vai um passo além do que muitos métodos baseados em IA localizando uma proteína no nível de célula única, e não como uma estimativa média em todas as células de um tipo específico. Essa localização de célula única pode identificar a localização de uma proteína em uma célula most cancers específica após o tratamento, por exemplo.
Os pesquisadores combinaram um modelo de linguagem de proteínas com um tipo especial de modelo de visão computacional para capturar detalhes ricos sobre uma proteína e célula. No remaining, o usuário recebe uma imagem de uma célula com uma porção destacada, indicando a previsão do modelo de onde a proteína está localizada. Como a localização de uma proteína é indicativa de seu standing funcional, essa técnica pode ajudar pesquisadores e médicos diagnosticar com mais eficiência doenças ou identificar metas de medicamentos, além de permitir que os biólogos entendam melhor como processos biológicos complexos estão relacionados à localização de proteínas.
“Você pode fazer esses experimentos de localização de proteínas em um computador sem precisar tocar em nenhum banco de laboratório, com esperança de economizar meses de esforço. Embora você ainda exact verificar a previsão, essa técnica pode agir como uma triagem inicial do que testar para o programa de biologia e co-líder da Yitong, um estudante de graduação do MIT.
A TSEO é acompanhada no artigo do co-líder, Xinyi Zhang, um estudante de graduação no Departamento de Engenharia Elétrica e Ciência da Computação (EECs) e o Centro de Eric e Wendy Schmidt no Broad Institute; Yunhao Bai do Broad Institute; e os autores seniores Fei Chen, professora assistente de Harvard e membro do Broad Institute, e Caroline Uhler, professora de engenharia de Andrew e Erna Viterbi em EECs e do MIT para o Methods, Methods e Sociedade (IDSs), que também é diretor de Wendy Methods e Wendy Methods (Wendy Methods e um pesquisador (IDSs). A pesquisa aparece hoje em Métodos da natureza.
Modelos colaboradores
Muitos modelos de previsão de proteínas existentes só podem fazer previsões com base nos dados de proteínas e celulares nos quais foram treinados ou não conseguem identificar a localização de uma proteína dentro de uma única célula.
Para superar essas limitações, os pesquisadores criaram um método de duas partes para a previsão da localização subcelular das proteínas invisíveis, chamada filhotes.
A primeira parte utiliza um modelo de sequência de proteínas para capturar as propriedades de determinação de localização de uma proteína e sua estrutura 3D com base na cadeia de aminoácidos que a forma.
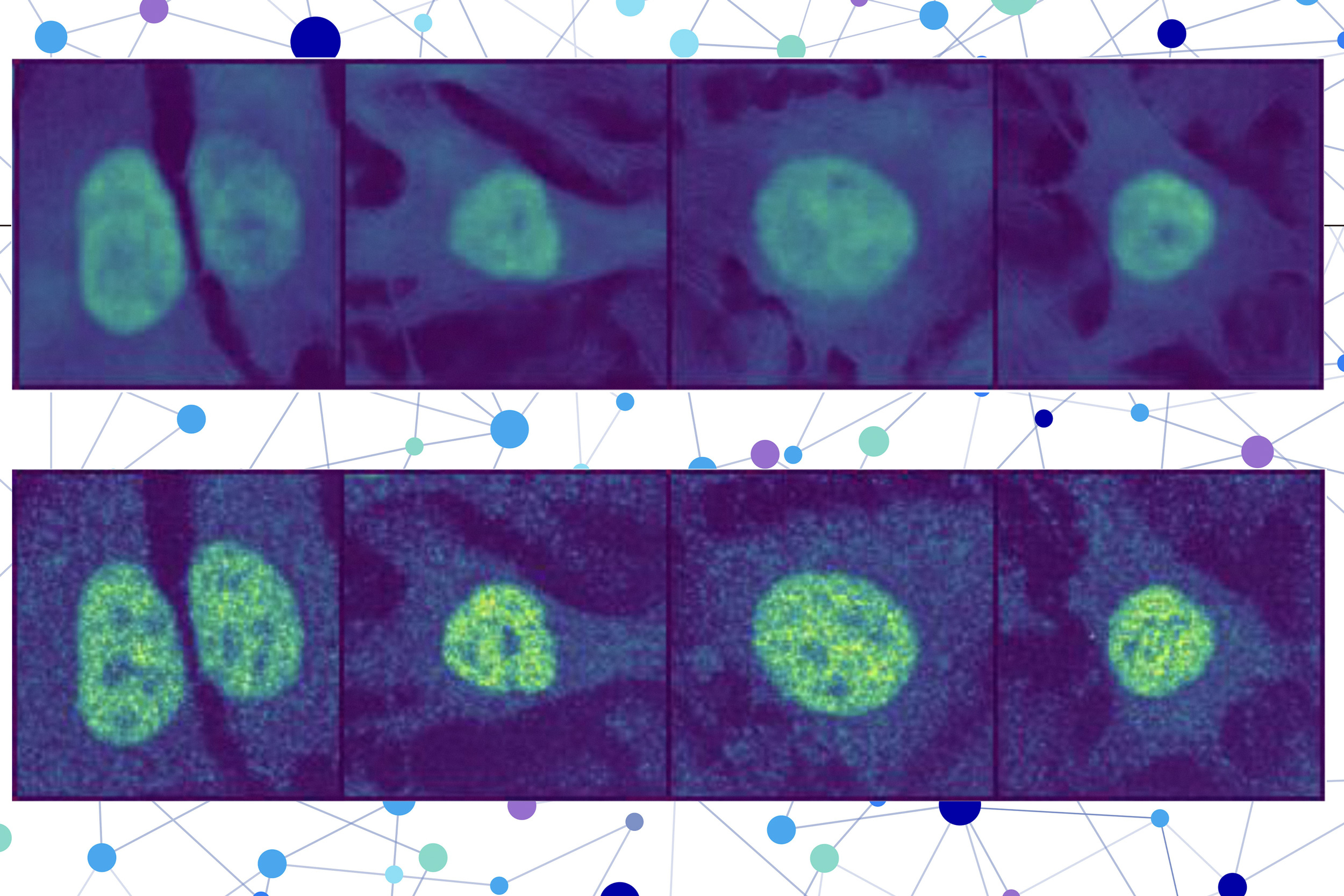
A segunda parte incorpora um modelo de pintura de imagem, projetado para preencher as partes ausentes de uma imagem. Esse modelo de visão computacional analisa três imagens manchadas de uma célula para coletar informações sobre o estado dessa célula, como seu tipo, recursos individuais e se está sob estresse.
Os filhotes se juntam às representações criadas por cada modelo para prever onde a proteína está localizada dentro de uma única célula, usando um decodificador de imagem para produzir uma imagem destacada que mostra o native previsto.
“Células diferentes dentro de uma linha celular exibem características diferentes, e nosso modelo é capaz de entender essa nuance”, diz Tseo.
Um usuário insere a sequência de aminoácidos que formam as imagens de proteínas e três células – uma para o núcleo, uma para os microtúbulos e outra para o retículo endoplasmático. Então os filhotes fazem o resto.
Um entendimento mais profundo
Os pesquisadores empregaram alguns truques durante o processo de treinamento para ensinar os filhotes a combinar informações de cada modelo de forma que possa fazer um palpite educado sobre a localização da proteína, mesmo que não tenha visto essa proteína antes.
Por exemplo, eles atribuem ao modelo uma tarefa secundária durante o treinamento: nomear explicitamente o compartimento da localização, como o núcleo celular. Isso é feito juntamente com a tarefa principal de pintura para ajudar o modelo a aprender com mais eficiência.
Uma boa analogia pode ser uma professora que pede aos alunos que desenhem todas as partes de uma flor, além de escrever seus nomes. Esta etapa additional foi encontrada para ajudar o modelo a melhorar sua compreensão geral dos possíveis compartimentos celulares.
Além disso, o fato de os filhotes serem treinados em proteínas e linhas celulares ao mesmo tempo ajuda a desenvolver uma compreensão mais profunda de onde em uma celular proteínas tendem a se localizar.
Os filhotes podem até entender, por si só, como diferentes partes da sequência de uma proteína contribuem separadamente para sua localização geral.
“A maioria dos outros métodos geralmente exige que você tenha uma mancha da proteína primeiro, para que você já o viu em seus dados de treinamento. Nossa abordagem é única, pois pode generalizar entre proteínas e linhas celulares ao mesmo tempo”, diz Zhang.
Como os filhotes podem generalizar para proteínas invisíveis, ele pode capturar alterações na localização impulsionadas por mutações proteicas únicas que não estão incluídas no atlas de proteínas humanas.
Os pesquisadores verificaram que os filhotes poderiam prever a localização subcelular de novas proteínas em linhas celulares invisíveis, conduzindo experimentos de laboratório e comparando os resultados. Além disso, quando comparado a um método de IA basal, os filhotes exibiram, em média, menos erro de previsão nas proteínas que testaram.
No futuro, os pesquisadores desejam aprimorar filhotes para que o modelo possa entender as interações proteína-proteína e fazer previsões de localização para múltiplas proteínas dentro de uma célula. A longo prazo, eles querem permitir que os filhotes façam previsões em termos de tecido humano vivo, em vez de células cultivadas.
Esta pesquisa é financiada pelo Eric e Wendy Schmidt Heart no Broad Institute, The Nationwide Institutes of Well being, The Nationwide Science Basis, The Burroughs Welcome Fund, The Searle Students Basis, The Harvard Stem Cell Institute, Merkin Institute, Workplace of Naval Analysis e o Departamento de Energia.