As proteínas são os cavalos de trabalho que mantêm nossas células funcionando e existem muitos milhares de tipos de proteínas em nossas células, cada uma desempenhando uma função especializada. Os pesquisadores sabem há muito tempo que a estrutura de uma proteína determina o que pode fazer. Mais recentemente, os pesquisadores estão apreciando que a localização de uma proteína também é crítica para sua função. As células estão cheias de compartimentos que ajudam a organizar seus muitos habitantes. Juntamente com as organelas conhecidas que adornam as páginas dos livros de biologia, esses espaços também incluem uma variedade de compartimentos dinâmicos sem membrana que concentram certas moléculas para executar funções compartilhadas. Saber onde uma determinada proteína se localiza e com quem co-localiza, pode, portanto, ser útil para uma melhor compreensão que a proteína e seu papel na célula saudável ou doente, mas os pesquisadores careciam de uma maneira sistemática de prever essas informações.
Enquanto isso, a estrutura da proteína foi estudada por mais de meio século, culminando na ferramenta de inteligência synthetic alfafold, que pode prever a estrutura de proteínas do código de aminoácidos de uma proteína, a corda linear de blocos de construção nela que se dobra para criar sua estrutura. Alphafold e modelos como se tornaram ferramentas amplamente usadas na pesquisa.
As proteínas também contêm regiões de aminoácidos que não se dobram em uma estrutura fixa, mas são importantes para ajudar as proteínas a unir compartimentos dinâmicos na célula. O professor Richard Younger e colegas do MIT se perguntaram se o código nessas regiões poderia ser usado para prever a localização de proteínas da mesma maneira que outras regiões são usadas para prever a estrutura. Outros pesquisadores descobriram algumas seqüências de proteínas que codificam a localização de proteínas e alguns começaram a desenvolver modelos preditivos para localização de proteínas. No entanto, os pesquisadores não sabiam se a localização de uma proteína em qualquer compartimento dinâmico poderia ser previsto com base em sua sequência, nem tinha uma ferramenta comparável ao alfafold para prever a localização.
Agora, jovem, também membro do Instituto Whitehead de Pesquisa Biológica; Younger Lab PostDoc Henry Kilgore; Regina Barzilay, professora distinta da Escola de Engenharia de IA e Saúde no Departamento de Engenharia Elétrica e Ciência da Computação do MIT e investigador principal no Laboratório de Ciência da Computação e Inteligência Synthetic (CSAIL); E os colegas criaram esse modelo, que eles chamam de ProtGps. Em um artigo publicado em 6 de fevereiro no diário Ciênciacom os primeiros autores Kilgore e os estudantes de pós-graduação do Barzilay Lab, Itamar Chinn, Peter Mikhael e Ilan Mitnikov, a equipe interdisciplinar estreia seu modelo. Os pesquisadores mostram que os protgps podem prever qual dos 12 tipos conhecidos de compartimentos que uma proteína se localizará, bem como se uma mutação associada à doença mudará essa localização. Além disso, a equipe de pesquisa desenvolveu um algoritmo generativo que pode projetar novas proteínas para se localizar em compartimentos específicos.
“Minha esperança é que este seja um primeiro passo em direção a uma plataforma poderosa que permite que as pessoas que estudam proteínas façam sua pesquisa”, diz Younger, “e que nos ajuda a entender como os humanos se desenvolvem nos organismos complexos que eles são, como as mutações perturbam aquelas processos naturais e como gerar hipóteses terapêuticas e projetar medicamentos para tratar a disfunção em uma célula. ”
Os pesquisadores também validaram muitas das previsões do modelo com testes experimentais nas células.
“Isso realmente me empolgou por poder ir do design computacional até experimentar essas coisas no laboratório”, diz Barzilay. “Existem muitos trabalhos interessantes nessa área de IA, mas 99,9 % deles nunca são testados em sistemas reais. Graças à nossa colaboração com o Younger Lab, fomos capazes de testar e realmente aprendemos o quão bem nosso algoritmo está indo. ”
Desenvolvendo o modelo
Os pesquisadores treinaram e testaram protgps em dois lotes de proteínas com localizações conhecidas. Eles descobriram que isso poderia prever corretamente onde as proteínas acabam com alta precisão. Os pesquisadores também testaram o quão bem os protgps poderiam prever alterações na localização de proteínas com base em mutações associadas à doença dentro de uma proteína. Muitas mutações – alterações na sequência de um gene e sua proteína correspondente – contribuíram ou causam doenças com base em estudos de associação, mas as maneiras pelas quais as mutações levam a sintomas da doença permanecem desconhecidas.
Descobrir o mecanismo de como uma mutação contribui para a doença é importante porque os pesquisadores podem desenvolver terapias para corrigir esse mecanismo, impedindo ou tratando a doença. Jovens e colegas suspeitavam que muitas mutações associadas à doença pudessem contribuir para a doença, mudando a localização de proteínas. Por exemplo, uma mutação pode tornar uma proteína incapaz de ingressar em um compartimento contendo parceiros essenciais.
Eles testaram essa hipótese alimentar protGOs com mais de 200.000 proteínas com mutações associadas à doença e, em seguida, pedindo para prever onde essas proteínas mutadas localizariam e mediriam quanto sua previsão mudou para uma determinada proteína da versão regular para a mutada. Uma grande mudança na previsão indica uma mudança provável na localização.
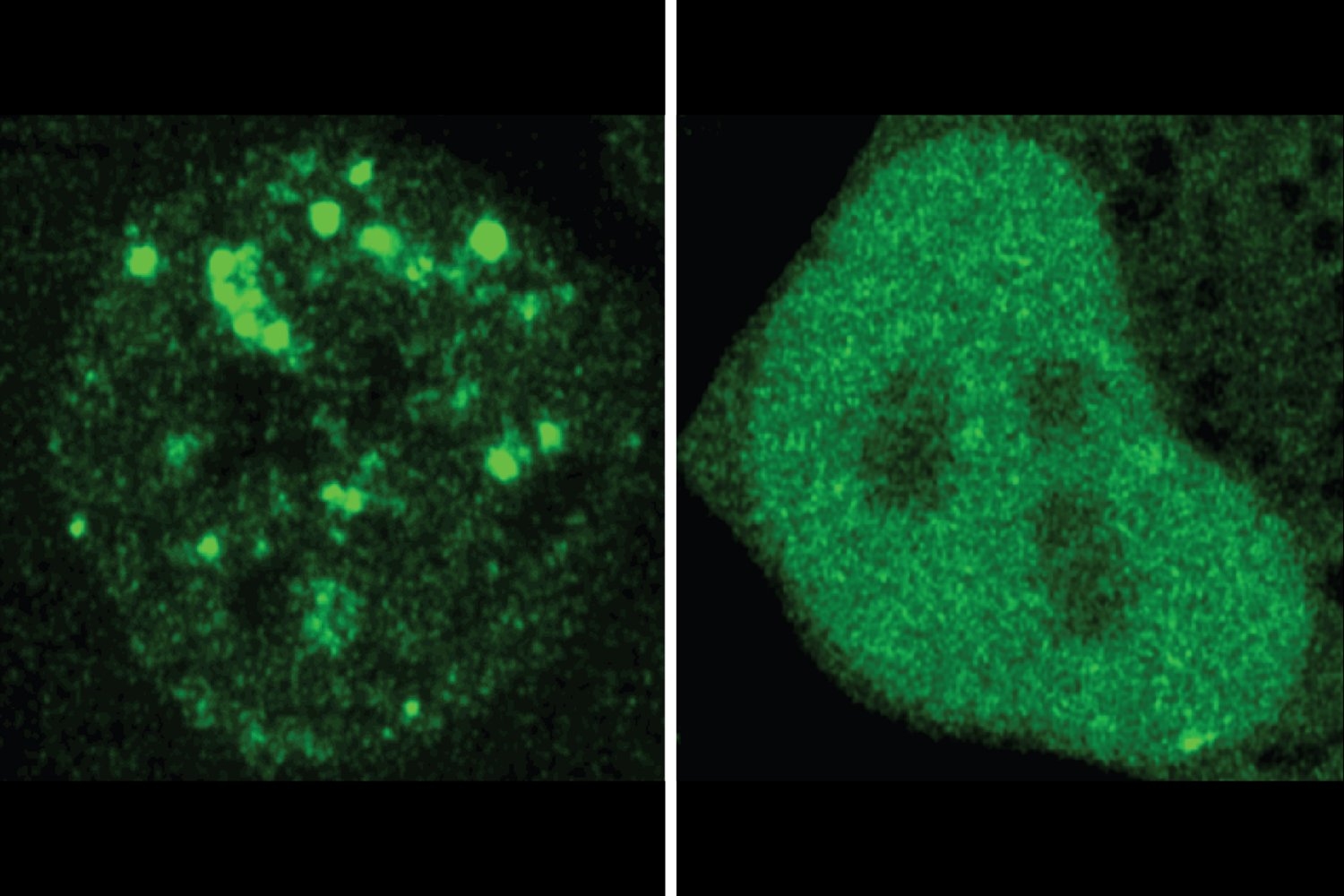
Os pesquisadores descobriram muitos casos em que uma mutação associada à doença parecia alterar a localização de uma proteína. Eles testaram 20 exemplos nas células, usando fluorescência para comparar onde, na célula, uma proteína regular e a versão mutada acabaram. Os experimentos confirmaram as previsões do ProTGPS. No whole, os achados apóiam a suspeita dos pesquisadores de que a localização incorreta pode ser um mecanismo de doença subestimado e demonstrar o valor do protgps como uma ferramenta para entender a doença e identificar novos caminhos terapêuticos.
“A célula é um sistema tão complicado, com tantos componentes e redes complexas de interações”, diz Mitnikov. “É tremendous interessante pensar que, com essa abordagem, podemos perturbar o sistema, ver o resultado disso e, assim, impulsionar a descoberta de mecanismos na célula ou até desenvolver terapêutica com base nisso”.
Os pesquisadores esperam que outros comecem a usar o ProtGPS da mesma maneira que usam modelos estruturais preditivos como o Alphafold, avançando vários projetos sobre função de proteína, disfunção e doença.
Indo além da previsão para a nova geração
Os pesquisadores ficaram empolgados com os possíveis usos de seu modelo de previsão, mas também queriam que seu modelo fosse além da previsão de localizações das proteínas existentes e permitisse que eles projetem proteínas completamente novas. O objetivo period que o modelo compensasse seqüências de aminoácidos totalmente novas que, quando formadas em uma célula, se localizavam em um native desejado. Gerar uma nova proteína que pode realmente cumprir uma função – neste caso, a função de localizar em um compartimento celular específico – é incrivelmente difícil. Para melhorar as probabilities de sucesso de seu modelo, os pesquisadores restringiram seu algoritmo a projetar apenas proteínas como as encontradas na natureza. Esta é uma abordagem comumente usada no design de medicamentos, por razões lógicas; A natureza teve bilhões de anos para descobrir quais sequências de proteínas funcionam bem e quais não.
Devido à colaboração com o Younger Lab, a equipe de aprendizado de máquina conseguiu testar se o gerador de proteínas funcionou. O modelo teve bons resultados. Em uma rodada, gerou 10 proteínas destinadas a se localizar no nucléolo. Quando os pesquisadores testaram essas proteínas na célula, descobriram que quatro deles se localizavam fortemente no nucléolo, e outros podem ter tido pequenos vieses em direção a esse native também.
“A colaboração entre nossos laboratórios tem sido tão generativa para todos nós”, diz Mikhael. “Aprendemos a falar as línguas um do outro, em nosso caso, aprendeu muito sobre como as células funcionam e, ao ter an opportunity de testar experimentalmente nosso modelo, conseguimos descobrir o que precisamos fazer para realmente fazer O modelo funciona e depois faz com que funcione melhor. ”
Ser capaz de gerar proteínas funcionais dessa maneira poderia melhorar a capacidade dos pesquisadores de desenvolver terapias. Por exemplo, se um medicamento deve interagir com um alvo que se localiza em um determinado compartimento, os pesquisadores poderiam usar esse modelo para projetar um medicamento para também localizar lá. Isso deve tornar o medicamento mais eficaz e diminuir os efeitos colaterais, pois o medicamento gastará mais tempo se envolvendo com seu alvo e menos tempo interagindo com outras moléculas, causando efeitos fora do alvo.
Os membros da equipe de aprendizado de máquina estão entusiasmados com a perspectiva de usar o que aprenderam com essa colaboração para projetar novas proteínas com outras funções além da localização, o que expandiria as possibilidades de design terapêutico e outras aplicações.
“Muitos trabalhos mostram que eles podem projetar uma proteína que pode ser expressa em uma célula, mas não que a proteína tenha uma função específica”, diz Chinn. “Na verdade, tínhamos design funcional de proteínas e uma taxa de sucesso relativamente enorme em comparação com outros modelos generativos. Isso é realmente emocionante para nós, e algo que gostaríamos de construir. ”
Todos os pesquisadores envolvidos veem o ProtGps como um começo emocionante. Eles prevêem que sua ferramenta será usada para aprender mais sobre os papéis da localização na função proteica e localização incorreta na doença. Além disso, eles estão interessados em expandir as previsões de localização do modelo para incluir mais tipos de compartimentos, testar mais hipóteses terapêuticas e projetar proteínas cada vez mais funcionais para terapias ou outras aplicações.
“Agora que sabemos que esse código de proteína para localização existe e que os modelos de aprendizado de máquina podem entender esse código e até criar proteínas funcionais usando sua lógica, que abre a porta para tantos estudos e aplicações em potencial”, diz Kilgore.