Uma vasta busca da diversidade pure levou os cientistas do McGovern Institute for Analysis do MIT e do Broad Institute of MIT e Harvard a descobrir sistemas antigos com potencial para expandir a caixa de ferramentas de edição do genoma.
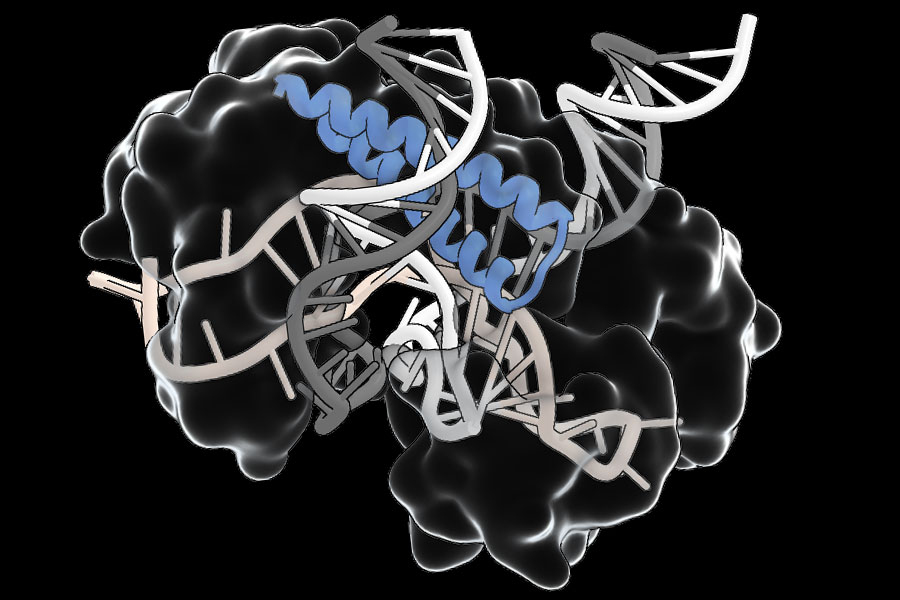
Esses sistemas, que os pesquisadores chamam de sistemas TIGR (RNA do guia intercalado em tandem), usam RNA para guiá -los para locais específicos no DNA. Os sistemas TIGR podem ser reprogramados para direcionar qualquer sequência de interesse de DNA e possuem módulos funcionais distintos que podem atuar no DNA direcionado. Além de sua modularidade, o TIGR é muito compacto em comparação com outros sistemas guiados por RNA, como o CRISPR, que é uma grande vantagem para entregá-lo em um contexto terapêutico.
Essas descobertas são Relatado on -line em 27 de fevereiro no diário Ciência.
“Este é um sistema guiado por RNA muito versátil, com muitas funcionalidades diversas”, diz Feng Zhang, professor de neurociência de James e Patricia Poitras no MIT, que liderou a pesquisa. As proteínas associadas ao TIGR (TAS) que a equipe de Zhang encontraram compartilham um componente característico de ligação ao RNA que interage com um guia de RNA que o direciona para um native específico no genoma. Alguns cortaram o DNA naquele native, usando um segmento adjacente de corte de DNA da proteína. Essa modularidade pode facilitar o desenvolvimento de ferramentas, permitindo que os pesquisadores trocassem novos recursos úteis em proteínas naturais do TAS.
“A natureza é incrível”, diz Zhang, que também é investigador do McGovern Institute e do Howard Hughes Medical Institute, um membro central do Broad Institute, professor de ciências cerebrais e cognitivas e engenharia biológica do MIT, e co-diretor da MYT. “Tem uma enorme quantidade de diversidade, e estamos explorando essa diversidade pure para encontrar novos mecanismos biológicos e aproveitá -los para diferentes aplicações para manipular processos biológicos”, diz ele. Anteriormente, a equipe de Zhang adaptou os sistemas bacterianos CRISPR em ferramentas de edição de genes que transformaram a biologia moderna. Sua equipe também encontrou uma variedade de proteínas programáveis, tanto dos sistemas CRISPR quanto além.
Em seu novo trabalho, para encontrar novos sistemas programáveis, a equipe começou zerando em uma característica estrutural da proteína CRISPR-CAS9 que se liga ao guia de RNA da enzima. Esse é um recurso essencial que tornou o CAS9 uma ferramenta tão poderosa: “Ser guiado por RNA facilita a reprograma, porque sabemos como o RNA se liga a outro DNA ou outro RNA”, explica Zhang. Sua equipe pesquisou centenas de milhões de proteínas biológicas com estruturas conhecidas ou previstas, procurando qualquer pessoa que compartilhasse um domínio semelhante. Para encontrar proteínas mais distantes relacionadas, eles usaram um processo iterativo: de Cas9, identificaram uma proteína chamada IS110, que anteriormente havia sido demonstrada por outros para se ligar ao RNA. Eles então se concentraram nas características estruturais do IS110 que permitem a ligação ao RNA e repetiram sua pesquisa.
Nesse ponto, a busca apareceu tantas proteínas relacionadas à distância que eles se juntaram à inteligência synthetic para entender a lista. “Quando você está fazendo mineração iterativa e profunda, os acertos resultantes podem ser tão diversos que são difíceis de analisar usando métodos filogenéticos padrão, que dependem da sequência conservada”, explica Guilhem Faure, um biólogo computacional no laboratório de Zhang. Com um modelo de linguagem grande de proteína, a equipe conseguiu agrupar as proteínas que haviam encontrado em grupos de acordo com suas prováveis relações evolutivas. Um grupo separado do restante, e seus membros foram particularmente intrigantes porque foram codificados por genes com sequências repetitivas regularmente espaçadas que lembram um componente essencial dos sistemas CRISPR. Estes eram os sistemas TIGR-TAS.
A equipe de Zhang descobriu mais de 20.000 proteínas TAS diferentes, ocorrendo principalmente em vírus infectados por bactérias. Sequências na região repetitiva de cada gene-suas matrizes tigradas-codificam uma guia de RNA que interage com a parte de ligação ao RNA da proteína. Em alguns, a região de ligação ao RNA é adjacente a uma parte de corte de DNA da proteína. Outros parecem se ligar a outras proteínas, o que sugere que eles podem ajudar a direcionar essas proteínas para alvos de DNA.
Zhang e sua equipe experimentaram dezenas de proteínas TAS, demonstrando que alguns podem ser programados para fazer cortes direcionados ao DNA nas células humanas. Enquanto pensam no desenvolvimento de sistemas TIGR-TAS em ferramentas programáveis, os pesquisadores são incentivados por recursos que podem tornar essas ferramentas particularmente flexíveis e precisas.
Eles observam que os sistemas CRISPR só podem ser direcionados para segmentos de DNA que são ladeados por motivos curtos conhecidos como PAMs (motivos adjacentes do Protospacer). As proteínas TIGR TAS, por outro lado, não têm esse requisito. “Isso significa teoricamente, qualquer native do genoma deve ser alvo”, diz Rhiannon Macrae, consultor científico. Os experimentos da equipe também mostram que os sistemas TIGR têm o que Faure chama de “sistema de dupla guia”, interagindo com os dois fios da hélice dupla de DNA para casa em suas sequências-alvo, o que deve garantir que elas agem apenas onde são direcionadas pelo seu guia de RNA. Além disso, as proteínas Tas são compactas – um quarto do tamanho Cas9, em média -, facilitando a entrega, o que poderia superar um grande obstáculo à implantação terapêutica de ferramentas de edição de genes.
Animado por sua descoberta, a equipe de Zhang está agora investigando o papel pure dos sistemas tigrados em vírus, bem como como eles podem ser adaptados para pesquisa ou terapêutica. Eles determinaram a estrutura molecular de uma das proteínas do TAS que encontraram para funcionar em células humanas e usarão essas informações para orientar seus esforços para torná -las mais eficientes. Além disso, eles observam conexões entre os sistemas TIGR-TAS e certas proteínas de processamento de RNA em células humanas. “Acho que há mais lá para estudar em termos do que alguns desses relacionamentos podem ser, e isso pode nos ajudar a entender melhor como esses sistemas são usados em humanos”, diz Zhang.
Este trabalho foi apoiado pela Helen Hay Whitney Basis, Howard Hughes Medical Institute, Okay. Lisa Yang e Hock E. Tan Middle for Molecular Therapeutics, Broad Institute Institute Programmable Therapeutics Reward Donors, Pershing Sq. Basis, William Ackman, Neri Oxman, a família Phillips, J. e P. Poitras e o Btras Charitent.